Image processing with Generative Adversarial Networks (GANs) with limited data availability
- Ansprechperson:
Moritz Böhland
- Projektgruppe:
Maschinelles Lernen für Hochdurchsatzverfahren und Mechatronik (ML4HOME)
- Förderung:
HGF
- Starttermin:
2018
Künstliche neuronale Netze werden für komplexe Segmentierungsaufgaben genutzt. Einer der Anwendungsfälle ist die Segmentierung von Zellkernen in menschlichem Gewebe. Die Segmentierung kann im Anschluss unter anderem zur Analyse von Schilddrüsentumoren und deren Klassifikation genutzt werden [1, 2].

Überwachte Methoden benötigen jedoch annotierte Trainingsdaten, die meistens manuell annotiert werden. Die manuelle Annotation ist zeitaufwendig, teuer und fehleranfällig. Zusätzlich dazu ist die Variabilität zwischen verschiedenen Annotatoren groß. Generative Adversarial Networks (GANs) können genutzt werden um die manuelle Annotation zu ersetzen. Hierzu werden synthetische ungepaarte Annotationen erzeugt. Im Anschluss wird mit diesen Annotationen und den vorhandenen Bildern ein GAN trainiert. Das GAN lernt mit Hilfe dieser Bilder die Transformation zwischen den Domänen. Nachdem das Training abgeschlossen ist, kann das GAN angewandt werden, um einen vollsynthetischen Datensatz zu erzeugen. In unserer Arbeit untersuchen wir den Einfluss der ungepaarten synthetischen Annotationen auf die Qualität der synthetischen Datensätze, die das GAN erzeugt [3]. Zusätzlich dazu wird die Synthese von großen 2D- und 3D-Daten untersucht. Diese sind im Besonderen wichtig bei der Analyse von biologischen und biomedizinischen Daten, sowie in der Materialwissenschaft.
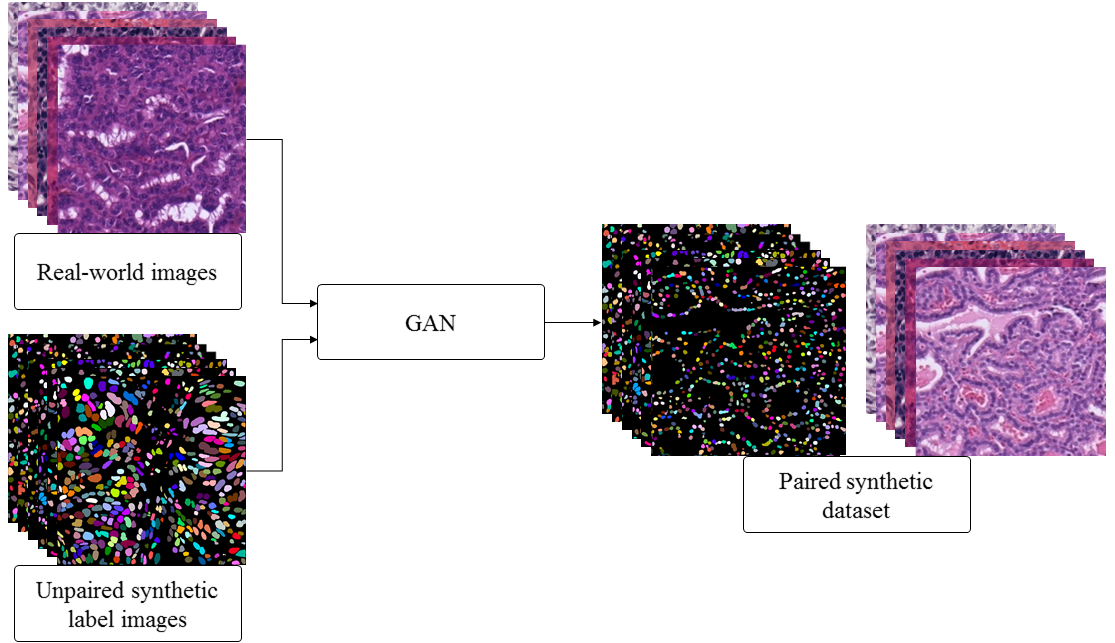
[1] M. Böhland, L. Tharun, et al.: “Machine learning methods for automated classification of tumors with papillary thyroid carcinoma-like nuclei: A quantitative analysis,” PLOS ONE, vol. 16, no. 9, pp. 1–21, 2021
[2] M. Böhland, O. Neumann, et al.: “Ciscnet - a Single-Branch Cell Nucleus Instance Segmentation and Classification Network,” in 2022 IEEE Int. Symp. Biomed. Imag. Challenges (ISBIC), 2022, pp. 1–5.
[3] M. Böhland, T. Scherr, et al.: Influence of synthetic label image object properties on GAN supported segmentation pipelines. In: Proceedings 29th Workshop Computational Intelligence, pp. 289–305 (2019)

