EMSIG – Event-driven Microscopy for Smart Microfluidic Single-cell Analysis
- Ansprechperson:
Prof. Dr.-Ing. Ralf Mikut, Angelo Jovin Yamachui Sitcheu, Nils Friederich, Oliver Neumann
- Projektgruppe:
- Förderung: HGF
- Projektbeteiligte:
FZJ
- Starttermin:
2023
- Endtermin:
2025
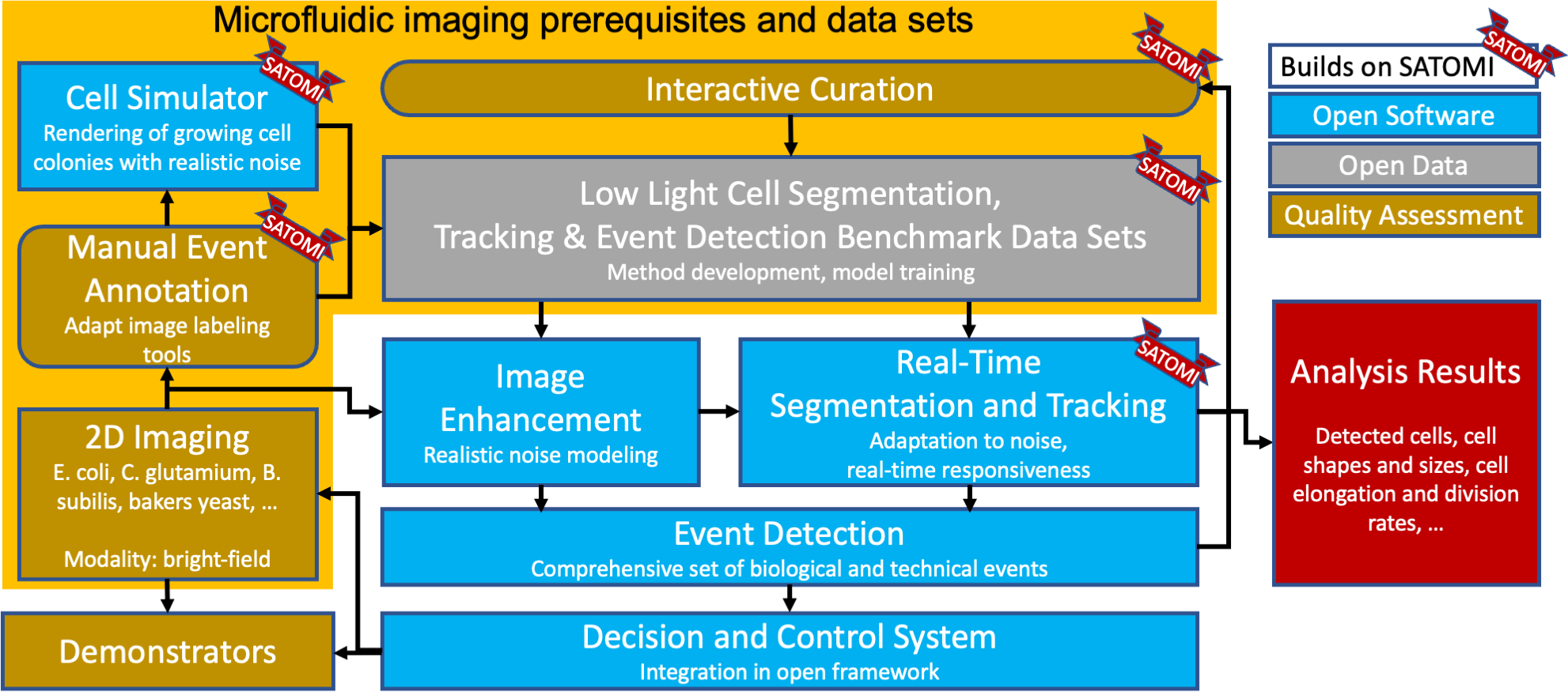
Die mikrofluidische Lebendzell-Mikroskopie (engl. MLCI: Microfluidic Live-Cell Imaging) ermöglicht es Biotechnologen, das räumlich-zeitliche Wachstum von Mikroben besser zu verstehen. Im Rahmen des „Helmholtz Imaging Platform (HIP)“-Projekts SATOMI wurden die auf Deep Learning basierenden Bildanalysewerkzeuge microbeSEG [1] und ObiWan-Microbi [2] entwickelt, um Offline-Analysen von hochqualitativen Daten durchzuführen. Diese Tools konzentrierten sich hauptsächlich auf die Herausforderungen der Segmentierung und Nachverfolgung von wachsenden Kolonien. Ein wichtiger Aspekt, der im Nachhinein festgestellt wurde, war das Fehlen einer kontinuierlichen Erfassung hochqualitativer Daten während der Experimente, die sich über Tage und Wochen erstreckten.
Das EMSIG-Projekt befasst sich mit diesem Problem, indem es dem MLCI Kapazitäten zur Erkennung von Live-Ereignissen zur Verfügung stellt. Ziel ist es, ein intelligentes und anpassungsfähiges ereignisbasiertes Erkennungs- und Steuerungssystem zu entwickeln, das vordefinierte biologische Ereignisse wie Zellwachstum und Zellteilung oder technische Ereignisse wie eine Verschlechterung der Bildqualität im Laufe der Zeit selbständig erkennt und entsprechende Echtzeitreaktionen auslöst. Zu diesen Reaktionen gehören zum Beispiel die Neufokussierung oder die Erhöhung der räumlich-zeitlichen Auflösung, um bestimmte Ereignisse zu beobachten.
Die Herausforderung bei EMSIG (Event-driven Microscopy for Smart mIcrofluidic sinGle-cell analysis) besteht darin, eine robuste und präzise Echtzeit-Klassifizierung von Ereignisauslösern an der Schnittstelle von Bildgebung und stochastischen biologischen Komponenten zu erreichen. Wenn dies gelingt, eröffnet dies völlig neue Erkenntnisse für die Charakterisierung schneller Prozesse, den Fortschritt bei der Automatisierung der Lebendzellmikroskopie und die Erhöhung des Technology Readiness Level (TRL) von MLCI.
[1] microbeSEG: A deep learning software tool with OMERO data management for efficient and accurate cell segmentation
[2] ObiWan-Microbi and microbeSEG: Deep Learning Tools for Microbial Image Analysis
Publikationen:
Friederich, N.; Yamachui Sitcheu, A. J.; Nassal, A.; Yildiz, E.; Pesch, M.; Beichter, M.; Scholtes, L.; Akbaba, B.; Lautenschlager, T.; Neumann, O.; Kohlheyer, D.; Scharr, H.; Seiffarth, J.; Nöh, K.; Mikut, R.
2025. at - Automatisierungstechnik, 73 (10), 808–823. doi:10.1515/auto-2025-0018
Yamachui Sitcheu, A. J.; Akbaba, B.; Beichter, M.; Friederich, N.; Kohlheyer, D.; Lautenschlager, T.; Mikut, R.; Nassal, A.; Noeh, K.; Pesch, M.; Pesch, M.; Scharr, H.; Scholtes, L.; Seiffarth, J.; Yildiz, E.
2025. Helmholtz Imaging Conference (2025), Potsdam, Deutschland, 25.–27. Juni 2025
Friederich, N.; Seiffarth, J.; Yamachui Sitcheu, A. J.; Yildiz, E.; Pesch, M.; Scholtes, L.; Neumann, O.; Scharr, H.; Nöh, K.; Mikut, R.
2024, Mai 14. 4th Helmholtz Imaging Conference (2024), Heidelberg, Deutschland, 14.–15. Mai 2024
Friederich, N.; Yamachui Sitcheu, A. J.; Nassal, A.; Pesch, M.; Yildiz, E.; Beichter, M.; Scholtes, L.; Akbaba, B.; Lautenschlager, T.; Neumann, O.; Kohlheyer, D.; Scharr, H.; Seiffarth, J.; Nöh, K.; Mikut, R.
2024. Proceedings : 34. Workshop Computational Intelligence: Berlin, 21.-22. November 2024. Ed.: H. Schulte, 169–192, KIT Scientific Publishing
Yamachui Sitcheu, A. J.; Seiffarth, J.; Friederich, N.; Yildiz, E.; Scherr, T.; Neumann, O.; Wollenhaupt, B.; Scharr, H.; Nöh, K.; Kohlheyer, D.; Mikut, R.
2023. Helmholtz Imaging Conference (2023), Hamburg, Deutschland, 14.–16. Juni 2023

